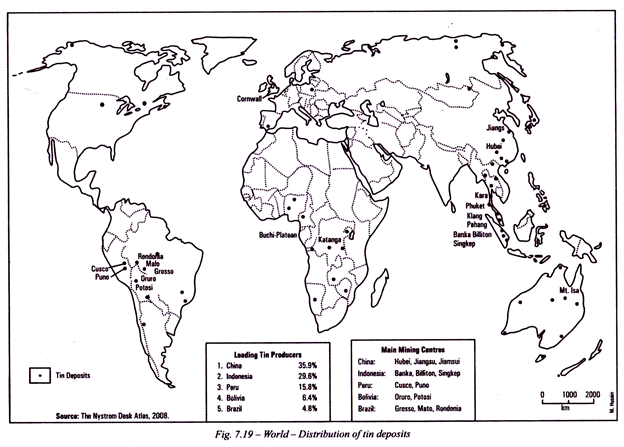
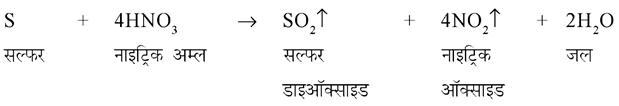
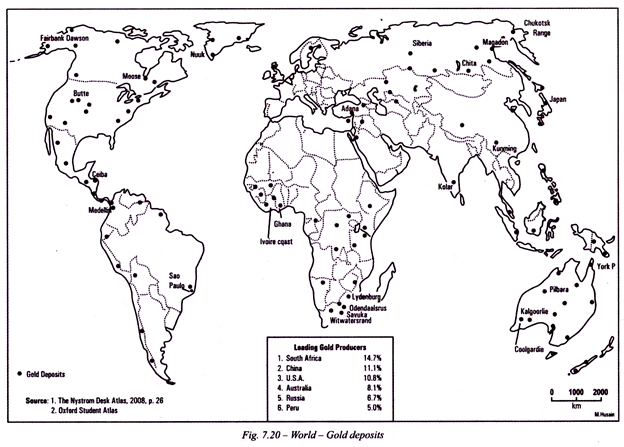
Read this article in Hindi to learn about:- 1. Type-I यूकैरियोटस ट्रांसक्रिप्शन Transcription in यूकैरियोटस राइबोसोमल आर.एन.ए. ट्रांसक्रिप्शन बाए आर.एन.ए. पॉलीमरेज-I (Type-I Eukaryotic Transcription in Eukaryotes Ribosomal RNA Transcription by RNA Polymerase- I) 2. अनुलेखन का प्रारम्भ व अनुलेखन कारक (Initiation of Transcription and Transcription Factors) and Other Details.
Type-I यूकैरियोटस ट्रांसक्रिप्शन Transcription in यूकैरियोटस राइबोसोमल आर.एन.ए. ट्रांसक्रिप्शन बाए आर.एन.ए. पॉलीमरेज-I (Type-I Eukaryotic Transcription in Eukaryotes Ribosomal RNA Transcription by RNA Polymerase- I):
RNA Polymerase केवल Ribosomal RNA के Gene को Transcription करता है । यह Transcription एक ही Promoter से प्रारम्भ होता है, जो मानव कोशिका में अध्ययन किया गया है ।
यह एक प्रमुख Promoter होता है, जो (UCE) Upstream Control Element से 700 Base Pair से अलग होता है । Core Promoter प्रारम्भ बिन्दु को घेरे होता है, जो -15 से +20 तक होता है तथा यह Transcription की क्रिया के लिए उपयुक्त होता है ।
RNA Polymerase को दो कारकों की आवश्यकता होती है:
ADVERTISEMENTS:
1. Upstream Binding Factor I (UBF-I),
2. Upstream Control Element (UCE) ।
एक और Factor की आवश्यकता होती है, जो चार Protein की इकाई से मिलकर बना होता है, जो UBF-I Factor से जुड़ता है । जब ये दोनों Factor जुड़ जाते हैं, तब RNA Poly-I भी Core Promoter से जुड़ जाता है तथा Transcription प्रारम्भ हो जाता है ।
m-RNA Transcription by RNA Polymerase-II:
ADVERTISEMENTS:
ऐसे Gene जो RNA Poly-II द्वारा Transcribe किये जाते हैं । वे अत्यधिक भिन्नता लिये होते हैं क्योंकि वे विभिन्न प्रकार के Protein को Code करते हैं और भिन्न-भिन्न समय पर कार्य करते हैं । Enzyme RNA Poly-II स्वतंत्र रूप से Transcription को प्रारम्भ नहीं कर सकता है ।
अत: ये अन्य Transcription कारकों पर निर्भर करता हैं । जिन्हें TF-II [Transcription Factor-II] RNA Poly-II के लिए Promoter Transcription Strand बिन्दु के 5′ Prime Site पर Present होता है । इन छोटी Sequence को Element कहा जाता है जो -25, -75 and -90(i) होते हैं ।
1. TATA Box Hogness Box की Sequence TATA AAT होती है, जो सभी Eukaryotes में पायी जाती है ।
2. -75 पर Present Sequence CAAT Box से दर्शायी जाती है ।
ADVERTISEMENTS:
3. GC Box, -90 पर Present होता है, जिसकी Sequence GGGCCGGG होती है ।
कुछ अन्य Sequence जिन्हें Enhancers भी कहा जाता है । इनकी कोई अपनी Promoter Activity नहीं होती है, परन्तु ये Transcription को Stimulate करते हैं । Enhancers अपनी क्रिया में निश्चिता दर्शाता है और इसलिए वे Gene Expression को नियंत्रित करते हैं ।
RNA Polymerase के Primary Transcribe को Heterogeneous Nucleus RNA (hn. RNA) कहते हैं । यह अधिक बड़े अणु हो सकते हैं, लगभग 10 Kilo Bases तक के, क्योंकि इनमें DNA में पाये जाने वाले सभी Entrons मौजूद होते है ।
Transfer RNA Transcription by RNA Polymerase III:
RNA Poly-III द्वारा जिस Gene को Transcribe किया जाता है (सभी 5.S rRNA, tRNA तथा कुछ छोटे Nucleus तथा Cytoplasmic RNA) । इनमें सामान्यतः 300 Nucleotide पाये जाते हैं । Entron अनुपस्थित होते हैं । कई छोटे tRNA Entron उपस्थित होते हैं । RNA Polymerase III के अलावा तीन Transcription Factor और पाये जाते हैं ।
जिन्हें TF III A, TF III B, TF III C कहते हैं । ये सभी 55S RNA के निर्माण के लिए आवश्यक होते हैं । इनमें से दो करक TF III aB, IF III C tRNA Gene के Transcription के लिए भी आवश्यक होते हैं और इसके बाद भी विधि m-RNA के समान ही होती हैं ।
Termination of Eukaryotic Transcription:
Termination की क्रियाविधि के बारे में कुछ बातें ही ज्ञात हैं । RNA Polymerase I, III की Termination की क्रियाएँ भिन्न होती है, परन्तु RNA Poly II के Termination के बारे में अब तक ज्ञात नहीं है । Termination Down Stream से 1000 Base Pair पहले 3′ Primer End पर होता है, जो Cleavage होता है ।
Termination में एक 18 Base Termination Sequence को पहचान जाता है । Polymerase-III के Base में Termination RNA Chain में Present Uracil पर Termination होता है । यह Uracil एक GC-Rich Region में Present होता है । सम्भवत: यह Region Enzyme की गति को धीमे कर देता है ।
प्रोकेरियोट् में mRNA का समापन (Termination of m-RNA in Prokaryotes):
DNA पर कुछ समापन संकेत पाए जाते हैं, जिनके द्वारा m-RNA श्रृंखला का समापन होता है । जीवाणुओं (Bacteria) में ये समापन संकेत (Termination Signals) एक अन्य प्रोटीन करक के द्वारा पहचाने जाते हैं ।
DNA क्रम जो समापन संकेत प्रदाय करते हैं, समापक (Terminator) कहलाते हैं । यह प्रोटीन कारक रहो (Rho,p) करक के नाम से जाना जाता है । इसका अणुभार 60,000 होता है, यह ई.कोलाई (E.Coli) से पृथक किया जाता है ।
यह कारक विट्रो (Vitro) में कम आयन शक्ति पर समापन करता है, लेकिन अन्य m-RNA के समापन के लिए आवश्यक नहीं होता है । रहो कारक (Rho Factor) के कार्य की क्रियाविधि ठीक से ज्ञात नहीं है, परन्तु इतना अवश्य ज्ञात है कि RNA पॉलिमेरेज एन्जाइम का भाग नहीं होता है ।
यूकेरियोट् में mRNA का समापन (Termination of m-RNA in Eukaryotes):
यूकैरियोट्स (Eukaryotes) में अनुलेखन के समय RNA पॉलिमेरेज-II का समापन स्थल के द्वारा प्रोकैरियोट्स (Prokaryotes) के समान होता है, लेकिन यह समापन स्थल, mRNA के 3′ सिरे के स्थल से दूर होता है । पश्च अनुलेखन विदलन के द्वार mRNA 3′ सिरा निर्मित होता है ।
यह विदलन ‘Snurp’ के द्वारा प्राप्त किया जाता है । ‘Snurp’ छोटा केन्द्रीय RNA प्रोटीन काम्प्लेक्स (Small Nuclear RNA-Protein Complex) पश्च अनुलेखन विदलन के लिए उपयोग होता है, जो कि अभी तक पहचाना नहीं गया है, लेकिन विघटित जीन्स में इन्ट्रान सप्लाइसिंग में उपयोग होता है ।
सारणी– तीन विभिन्न प्रकार के RNAPs के गुण (Properties of Three Kinds of RNAPs):
अनुलेखन के प्रारम्भ में प्रमोटर, एन्हांसर व साइलेन्सर स्थलों की भूमिका (Role of Promoter, Enhancer and Silencer Sites for Initiation of Transcription):
यूकैरियोट्स में प्री-आर-आर एन.ए. (Pre r-RNA) का निर्माण RNAP I से, प्री-एम-आर.एन.ए. (Pre m-RNA), हेटेरोजेनस न्यूक्लियर आर.एन.ए (Heterogeneous Nuclear RNA-hn RNA) व अनेक Sn RNAs का निर्माण RNAP II से साथ t-RNAs, 5S RNA, U6 Sn RNA का निर्माण RNAP II की मदद से होता है । विभिन्न प्रकार के RNAPs के लिए भिन्न-भिन्न प्रकार के प्रमोटर क्रमों (Promoter Sequences) की पहचान की गई है ।
(a) आर. एन. ए. पोलीमेरेज I के प्रमोटर्स (Promoter of RNAP I):
आर. एन. ए. पोलीमेरेज I (RNAP I) के प्रमोटर्स में कम से कम दो प्रमुख घटक होते हैं:
(i) अपस्ट्रीम कंट्रोल एलीमेंट (Upstream Control Elements) जिन्हें, संक्षिप्त नाम UCE से सम्बोधित किया जाता है । GC क्षारकों से समृद्ध UCE (-180 से -170) अपस्ट्रीम दिशा में स्थित होते हैं ।
(ii) कोर क्षेत्र (Core Regions) -45 से +20 के बीच स्थित यह क्षेत्र अनुलेखन प्रारम्भ बिन्दु (Transcription Start Site) को ओवरलैप करता है ।
(b) आर. एन. ए. पोलीमेरेज II के प्रमोटर्स, एन्हांसर व साइलेंसर स्थल (Promoter, Enhancer and Silencer Sites of RNAP II):
यूकैरियोटिक RNAP II के प्रमोटर क्रमों में आधारी (मूल) अनुलेखन (Basal Transcription) के लिए कोर प्रमोटर्स तथा एक्टीवेटर पर निर्भर अनुलेखन के लिए कुछ अन्य नियंत्रक क्रम (Regulatory Sequences) होते हैं ।
कोर प्रमोटर (Core Promoter):
40bp तक लम्बा होता है । यह क्रम बेसल ट्रांसक्रिप्शन फैक्टर (Basal Transcription Factors) जिन्हें जनरल ट्रांसक्रिप्शन फैक्टर (General Transcription Factor (GTFs)) भी कहा जाता है, की मदद से RNAP II द्वारा अनुलेखन को सही दिशा में सम्पादित कराने में सहायक होता है ।
विभिन्न प्रकार के कोर प्रमोटर्स, जिनका अध्ययन किया जा चुका है, निम्नलिखित हैं:
(i) टाटा बॉक्स (TATA Box):
A व T क्षारकों के समृद्ध होने के कारण ही, इसे TATA बॉक्स कहा जाता है । यह अनुलेखन प्रारम्भ स्थल (Transcription Start Site) से -30bp अपस्ट्रीम स्थित होता है । यह TF II D की TATA बाइण्डिंग प्रोटीन्स (TBP) से जुड़ता है । यह 7bp लम्बा होता है । इसका क्रम प्रायः TATAATAAT होता है । यह GC आधिक्य क्षेत्र से घिरा रहता है ।
(ii) बी. आर. ई. (BRE) (TF II B Recognition Element):
यह टाटा बॉक्स के तुरन्त बाद अपस्ट्रीम दिशा में स्थित होता है तथा TF II B की ओर प्रमोटर्स के प्रति बन्धुता (Affinity) बढ़ा देता है । इसी कारण इसे TF II B पहचान तत्व (TF II B Recognition Element) भी कहते हैं अर्थात् B Recognition Element ।
(iii) Inr (Initiator):
आरम्भ स्थल (Start Site) को धारण करने वाला यह एक संरक्षित क्रम है । इसका कार्य स्वयं या फिर TATA या DPE संरचनाओं की मदद से अनुलेखन के सही प्रारम्भ को निर्देशित करना हैं ।
(iv) डाउनस्ट्रीम प्रमोटर एलीमेण्ट (Downstream Promoter Element-DPE):
यह भी एक संरक्षित क्रम है जो प्रारम्भ बिन्दु से -30bp डाउन स्ट्रीम स्थित होता है । एक प्रारूपिक DPE वाले प्रमोटर में Inr व DPE संरचनाएँ होती हैं, लेकिन TATA बॉक्स का अभाव होता है ।
TF II D अपनी TAF इकाइयों द्वारा Inr व DPF संरचनाओं से जुड़ जाता है । आप जान चुके हैं कि सभी प्रमोटर्स में TATA बॉक्स की उपस्थित अनिवार्य नहीं तथा TATA बॉक्स की कमी की भरपाई DPE द्वारा हो जाती है ।
अत: यूकैरियोटिक प्रमोटर्स को मुख्यतः दो वर्गों में बाँटा जा सकता है- टाटा चालित प्रमोटर्स (TATA Driven Promoters) तथा डी पी ई चालित प्रमोटर्स (DPE Driven Promoters) ।
TATA बॉक्स की तुलना प्रोकैरियोट्स के प्रेबनाउ (Prebnow) बॉक्स से की जाती है । TATA बॉक्स से अपस्ट्रीम दिशा में -60 या -100bp दूरी पर GC बॉक्स तथा -80bp दूरी पर CAAT बॉक्स स्थित होता है । CAAT बॉक्स अनुलेखन की शुरुआत करने के लिए आवश्यक होता है ।
इसका क्रम प्राय: निम्न प्रकार से होता है:
इसी प्रकार GC बॉक्स कई प्रतियों में मौजूद रहता है तथा यह CAAT बॉक्स के साथ मिलकर अनुलेखन की क्षमता का निर्धारण रहता है । इसका क्रम GGGCGG होता है ।
यूकैरियोटिक प्रमोटर्स में प्रायः ऐसे भी घटक स्थित होते हैं, जो आर. एन. ए. पोलीमेरेज के अतिरिक्त अन्य प्रोटीन्स से भी अन्योन्य क्रिया करते हैं । प्रारम्भ बिन्दु से 100 से 200bp अपस्ट्रीम स्थित इन घटकों को एन्हांसर्स (Enhancers) या अपस्ट्रीम एक्टीवेशन साइट्स (Upstream Activation Sites, UAS) कहा जाता है ।
इनकी विशेषता है कि सैकडों-हजारों क्षारक युग्म (bp) अपस्ट्रीम या डाउन स्ट्रीम खिसक जाने पर भी इनके कार्य पर प्रभाव नहीं पड़ता । एन्हांसर का हिन्दी अर्थ है आवर्धक अर्थात् बढ़ा देने वाला । यह एन्हांसर किसी जीन के अनुलेखन की दर को 200 गुना तक बढ़ा देते हैं ।
अनुलेखन का नियंत्रण करने वाले एक प्रकार के तत्व साइलेंसर (Silencer) हैं, जो जीन अभिव्यक्ति का संदमन (Repression) करते हैं । एन्हांसर्स की तरह यह भी डी. एन. ए. के विशिष्ट स्थान है, जो प्रोटीन में अन्योन्य क्रिया (Interaction) करते हैं ।
(c) आर. एन. ए. पोलीमरेज- III के प्रमोटर्स (Promoters of RNAP III):
RNAP III के प्रमोटर्स के तीन समूहों की पहचान की जा चुकी है:
(i) t-RNA की जीन के प्रमोटर्स अनुलेखन प्रारम्भ स्थल (Transcription Start Site) से डाउनस्ट्रीम स्थित होते हैं । ये प्रमोटर्स दो घटकों से मिलकर बने होते हैं, जिनमें से प्रत्येक 10bp लम्बा होता है तथा यह एक दूसरे से 30-120bp दूरी पर स्थित होते हैं । इनको बॉक्स A व बॉक्स B नाम दिया गया है ।
(ii) मनुष्य के U6 sn-RNA जीन के प्रमोटर के घटक RNAP II के समान ही होते हैं । अर्थात् इनके प्रमोटर्स में TATA बॉक्स, PSE व OCT जैसी रचनाएँ पायी जाती है । इस जीन में अन्त: जीनी प्रमोटर घटक (Intragenic Promoter Elements) का अभाव होता है ।
(iii) 5s RNA जीन प्रमोटर्स में प्रारम्भ बिन्दु से 50bp डाउनस्ट्रीम, इंट्राजीनिक कण्ट्रोल क्षेत्र होते हैं । अनुलेखन इकाई में ही स्थित इस कण्ट्रोल क्षेत्र को बॉक्स C कहा जाता है । लेकिन विभिन्न जीनों में आर. एन. ए. पोलीमेरेज III के डाउनस्ट्रीम प्रमोटर्स की स्थिति भिन्न-भिन्न होती है ।
अनुलेखन का प्रारम्भ व अनुलेखन कारक (Initiation of Transcription and Transcription Factors):
अनुलेखन (अर्थात् RNA के संश्लेषण) के प्रारम्भ में अनेक अनुलेखन कारक (Transcription Factors) महत्वपूर्ण भूमिका निभाते हैं । TF II D या इसका एक भाग सभी प्रकार के RNAPs की क्रिया के सम्पादन के लिए आवश्यक होता है ।
लेकिन इसके अतिरिक्त अलग-अलग प्रकार के RNAPs के लिए अलग-अलग प्रकार के अनुलेखन कारकों की आवश्यकता होती है । यह नियम सभी यूकैरियोट्स के लिए समान है । वास्तव में अनुलेखन करक या ट्रांसक्रिप्शन फैक्टर्स वह प्रोटीन हैं, जो अनुलेखन के प्रारम्भ के लिए आवश्यक होती है ।
यहाँ यह उल्लेखनीय है कि यह प्रोटीन्स, RNAP का भाग नहीं होती । प्रायः जीनों की अभिव्यक्ति का नियमन एक्टीवेटर्स द्वारा होता है । इसीलिए इसे एक्टीवेटर डिपेन्डेंट ट्रांसक्रिप्शन (Activator Dependent Transcription) कहा जाता है ।
सभी जीनों में अनुलेखन को प्रारम्भ करने का कोई एक तरीका नही होता, विभिन्न प्रकार की जीनों में अनुलेखन की शुरुआत विभिन्न तरीकों से होती है । लेकिन सभी प्रकार के अनुलेखन में अनेक अनुलेखन कारक, प्रारम्भ स्थल (Start Point) से अपस्ट्रीम दिशा में स्थित विशिष्ट डी. एन. ए. क्रमों से अन्योन्य क्रिया (Interaction) करते हैं ।
यह अनुलेखन कारक डी. एन. ए. से RNAP के बन्धन (Binding) में मदद करते हैं । RNAP के DNA के साथ जुड़ जाने पर पूर्व प्रारम्भ कॉम्प्लैक्स (Prinitiation Complex) का निर्माण करते है । इसे अनुलेखन कॉम्प्लैक्स (Transcription Complex) भी कहा जाता है ।
इस कॉम्प्लैक्स के बनने के बाद ही अनुलेखन प्रारम्भ हो जाता है । यहाँ यह उल्लेखनीय है कि सभी प्रकार के अनुलेखन करक, डी. एन. ए. क्रमों को पहचान सकने में सक्षम नहीं होते । एक अनुलेखन कारक दूसरे अनुलेखन कारक को या RNAP को भी पहचान सकता है । अर्थात् उनसे अन्योन्य क्रिया (Interaction) कर सकता है ।
सामान्यतः RNAP I व RNAP III के डी. एन. ए. से जुड़कर अनुलेखन कॉम्प्लैक्स बनाने की क्रिया को तीन पदों में विभाजित किया जा सकता है, लेकिन RNAP II में यह क्रिया 4 पदों में पूर्ण होती है:
(i) न्यूक्लिएशन (Nucleation)- प्रमोटर क्रम की पहचान तथा उससे अनुलेखन कारक का जुड़ना (Binding) इस क्रिया का पहला पद बनाते है । इसे न्यूक्लिएशन नाम दिया गया है ।
(ii) दूसरे पद में प्रारम्भिक न्यूक्लिएशन के बाद कारक, RNAP को प्रमोटर स्थल तक पहुंचने में मदद करता है ।
(iii) RNAP का प्रवेश ।
(iv) कॉम्प्लैक्स का परिपक्वन (Complex Maturation)- केवल RNAP II के लिए ।
सारणी- विभिन्न प्रकार के अनुलेखन कारक व उनसे सम्बन्धित जीन व RNAPs (Various Transcriptional Factors and their Genes and RNAPs):
आर. एन. ए. पोलिमरेज II (RNAP II) के अनुलेखन कॉम्प्लैक्स का निर्माण (Formation of Transcription Complex of RNAP II):
संघटक जीनों (Constitutive Genes), जिनके उत्पाद की कोशिका को हर समय आवश्यकता होती है । इस प्रकार के जीनों की अभिव्यक्ति ऊतक विशिष्ट व उद्दीपन विशिष्ट जीनों की अभिव्यक्ति के समान नियंत्रित नहीं की जाती ।
इस प्रकार के जेनेरिक प्रमोटर पर RNAP II द्वारा अनुलेखन का आरम्भ मूलभूत या आधारी अनुलेखन (Basal Transcription) कहलाता है । इसके लिए अनेक सामान्य अनुलेखन कारकों की आवश्यकता होती है ।
इन्हें जनरल ट्रांसक्रिप्शन फैक्टर (General Transcription Factor or GTFs) नाम दिया गया है, जो निम्न प्रकार कार्य करते हैं:
सारणी– प्रारम्भन कारक के रूप में यीस्ट, चूहे व मनुष्य में प्रयुक्त RNAPs II के अनुलेखन कारक (Transcriptional Factors of RNAPS II used as Initiation Factors in Yeast, Rat and Human):
अनुलेखन कारकों में डी. एन. ए. बाइण्डिंग (DNA Binding) व अनुलेखन सक्रियण (Transcription Activation) संरचनाएँ अलग-अलग होती हैं । जीन अभिव्यक्ति के नियमन के लिए यह करक अपने डी. एन. ए. बाइण्डिंग संरचनाओं द्वारा डी. एन. ए. के विशिष्ट क्रमों की पहचान करते हैं तथा डी. एन. ए. से जुड़ने (Binding) के बाद यह आर. एन. ए. पोलीमरेज या अन्य अनुलेखन कारकों के साथ अन्योन्य क्रिया करते हैं । ट्रांसक्रिप्शन एक्टीवेशन संरचनाएँ, अनुलेखन के सक्रियण के लिए प्रोटीन-प्रोटीन अन्योन्य क्रियाओं (Protein-Protein Interactions) में भाग लेती है ।
डी. एन. ए. बाइण्डिंग संरचनाएँ (DNA Binding Domains):
प्रोटीन्स में ऐसी अनेक संरचनाएँ होती हैं, जो डी. एन. ए. से अन्योन्य क्रिया द्वारा अनुलेखन का नियमन करती हैं ।
इनका संक्षिप्त वर्णन निम्नलिखित है:
(a) स्टीरॉइड रीसेप्टर्स (Steroid Receptors):
अनेक स्टीरॉइड रीसेप्टर्स अपने से मेल वाले स्टीरॉइड से सक्रिय होकर अन्त:कोशिकीय संग्राही (Intracellular Receptor) का सक्रियण कर देते हैं । अन्त:कोशिकीय संग्राही विशिष्ट DNA क्रमों (Sequences) से जुड़कर अनुलेखन प्रारम्भ कर देते हैं ।
स्टीरॉइड हॉर्मोन्स, रेटीनॉइड्स (Retinoids), विटामिन D व अन्य अनेक प्रकार के यौगिक स्टीरॉइड रिसेप्टर के सक्रिय कर सकते हैं । इस प्रकार की प्रोटीन्स (अर्थात् स्टीरॉइड रिसेप्टर) में हॉर्मोन के जुड़ने, डी. एन. ए. के जुड़ने व अनुलेखन सक्रियण के लिए अलग-अलग स्थान होते हैं । इसकी डी. एन. ए. बाइण्डिंग संरचना में 70 अमीनो अम्ल होते हैं, जिनमें से 8 संरक्षित सिस्टीन अवशेष दो जिंग प्रवर्धों (Zinc Fingers) का निर्माण करते हैं ।
(b) जिंक प्रोटीन्स (Zinc Proteins):
डी. एन. ए. बन्धन संरचना बनाने वाले जिंग प्रवर्ध (Zinc Fingers) की सबसे पहले पहचान RNAP III द्वारा 5S r-RNA जीन के अनुलेखन के लिए आवश्यक अनुलेखन कारक TF III A में की गई ।
जिंक प्रोटीन्स वास्तव में ऐसी प्रोटीन्स हैं, जिनमें जिंग फिंगर (Zinc Fingers), जिंक क्लस्टर्स (Zinc Clusters) व जिंक ट्विस्टस (Zinc Twists) जैसी संरचनाएँ पायी जाती है ।
इनमें से कुछ प्रोटीन्स में स्पष्ट डी. एन. ए. बाइण्डिंग संरचनाएँ पायी जाती हैं । ऐसी एक रचना में अमीनो अम्लों से बना एक लूप, जिसमें जिंक भी होता है बाहर की ओर निकलकर जिंग प्रवर्ध (Zinc Finger) का निर्माण करता है ।
कुछ अन्य प्रोटीन्स में जिंक क्लस्टर व जिंक कुण्डलन (Zinc Twist) जैसी रचना होती है, लेकिन जिंक प्रवर्ध (Zinc Fingers) की तुलना में जिंक क्लस्टर व जिंक कुण्डलन (Zinc Twists) के बारे में अधिक जानकारी उपलब्ध नहीं है ।
निम्न दो प्रकार की जिंक फिंगर्स का वर्णन किया गया है:
(i) Cys2/His2 प्रवर्ध (फिंगर):
इस प्रकार का प्रत्येक प्रवर्ध (Finger) लगभग 23 अमीनो अम्लों से बना होता है तथा दूसरे प्रवर्ध से 7-8 अमीनो अम्ल द्वारा जुड़ा होता है ।
इस प्रकार की फिंगर में निम्न कान्सेन्सस क्रम होता है:
Cys – X2 – Cys – X3 – Phe – X5 – Leu – X2 – His – X3 – His
जिंक प्रवर्ध ही डी. एन. ए. बाइण्डिंग में मदद करते हैं । TF III A में 9 जिंक फिंगर्स तथा ADR I में दो जिंक फिंगर्स पायी जाती हैं । अर्थात् ADR I में प्रोटीन का केवल एक छोटा-सा हिस्सा जिंक फिंगर्स बनाने में भाग लेता है जबकि TF III A में यह पूरी प्रोटीन में फैली रहती है ।
(ii) Cys2/Cys2 प्रवर्ध (फिंगर):
Cys2/Cys2 फिंगर्स वाली कुछ प्रोटीन्स, स्टीरॉइड रिसेप्टर्स (Steroid Receptors) होती हैं । ऐसे उत्परिवर्तन (Mutation) जो प्रोटीन के फिंगर क्षेत्र के अमीनो अम्लों में बदलाव ला देते हैं, इन प्रोटीन की डी. एन. ए. बाइण्डिंग क्षमता को भी समाप्त कर देते हैं । यह इस बात का स्पष्ट प्रमाण है कि जिंक फिंगर ही डी. एन. ए. बाइण्डिंग में भाग लेती है ।
सारणी- Cys2/His2 जिंक फिंगर से युक्त अनुलेखन कारक व अन्य प्रोटीन्स (Transcriptional Factors and Other Proteins Attached with Cys2/His2 Zine Finger):
सारणी- Cys2/His2 जिंक फिंगर से युक्त रेगुलेटरी प्रोटीन्स (Regulatory Proteins Attached with Cys2/Cys2 Zinc Finger):
(c) हैलिक्स टर्न हैलिक्स (Helix Turn Helix):
हैलिक्स टर्न हैलिक्स रचना की पहचान सबसे पहले जीवाणुभोजी λ (लैम्ब्डा) के रिप्रेसर्स में की कई थी, लेकिन आज ड्रोसोफिला व स्तनधारियों के अनेकानेक अनुलेखन कारकों में इनकी खोज की जा चुकी है ।
इनमें से एक α – हैलिक्स, डी. एन. ए. की मुख्य खाँच (Major Groove) में तथा दूसरी डी. एन. ए. से एक कोण बनाती हुई स्थित होती है । इसके लिए संक्षिप्ताक्षर नाम HTH प्रयोग किया जाता है ।
HTH, 20 अमीनो अम्लों से बना खण्ड है जिसमें 1-8 रेसीड्यु से बनी एक α – हैलिक्स, 8-11 रेसीड्यु से बना एक टर्न व 12-20 रेसीड्यु से बनी एक दूसरी α – हैलिक्स शामिल होती है । प्रोकैरियोट्स की CAP, λ Cro व Rep. जैसी डी. एन. ए. बाइण्डिंग प्रोटीन में हैलिक्स टर्न हैलिक्स (HTH) प्रकार की रचनाएँ पायी जाती है ।
(d) हैलिक्स-लूप-हैलिक्स (Helix-Loop-Helix-HLH):
इस वर्ग की प्रोटीन ल्यूसीन जिपर (Leucine Zipper) संरचनाओं से समानता प्रदर्शित करती है । ल्यूसीन जिपर प्रोटीन्स ही की तरह इनमें (HLH) में एक क्षारीय (Basic) क्षेत्र छोटा है, जो DNA से सम्पर्क बनाता है ।
इस क्षेत्र के पास वाला एक क्षेत्र डाइमर (Dimer) निर्माण में सहायक होता है । डाइमर निर्माण से सम्बन्धित क्षेत्र में तीन प्रमुख भाग होते है- एक हैलिक्स, एक लूप व एक दूसरी α हैलिक्स ।
HLH प्रोटीन्स की क्रिया क नियमन हेटेरोडाइमर (Heterodimer) के निर्माण द्वारा होता है तथा यह भिन्नत (Differentiation) व विकस (Development) में महत्वपूर्ण भूमिका निभाती है ।
Myo D प्रोटीन इसी प्रकार की प्रोटीन का एक उदाहरण है जो EzA प्रोटीन के साथ हेटेरोडाइमर बनाती है तथा पेशी कोशिकाओं के भिन्नत (Differentiation) में मदद करती हैं ।
(e) ल्यूसीन जिपर (Leucine Zipper):
स्तनधारियों के एक विषाणु SV40 के एक जीन के प्रमोटर में 5 बार पुनरावृत्त करने वाली संरचना से जुड़ने वाली प्रोटीन SPI की खोज रॉबर्ट टिजान (Rober Tijan) ने 1984 में की । यह प्रोटीन इस जीन क चयनात्मक रूप से सक्रियण करने में सक्षम पायी गई । यूकैरियोट्स में C/EBP इसी प्रकार की एक प्रोटीन है । यह प्रोटीन CAAT से जुड़ती है ।
ल्यूसीन जिपर (Leucine Zipper) के पास क्षारीय (Basic) अमीनो अम्ल आर्जीनिन (Arginine) व लाइसिन (Lysine) (arg/lys) पाये जाते हैं, जो डी. एन. ए. बाइण्डिंग में मदद करते हैं । दो अणुओं के बीच जिपर निर्माण (Zippering) आर्जीनीन व लाइसिन को ऐसी स्थिति में ला देती है जिससे डी. एन. ए. से इसका संयोजन दोनों की समान सममिति के करण आसान हो जाता है । जिपर निर्माण (Zippering) के फलस्वरूप Y के आकार के डाइमर का निर्माण हो जाता है ।
अनुलेखन एक्टीवेटर्स व को-एक्टीवेटर्स (Transcription Activators and Co-Activators):
यूकैरियोट्स में अनुलेखन एक जटिल प्रक्रिया है तथा ट्रांसक्रिप्शन इनीशिएशन कॉम्प्लैक्स के संयोजन में भी अनेकानेक प्रकार की प्रोटीन डी. एन. ए. व प्रोटीन-प्रोटीन अन्योन्य क्रियाएँ (Interactions) भाग लेती है ।
अनुलेखन के इनीशिएशन कॉम्प्लैक्स के संयोजन में निम्नांकित प्रकार की समस्याएँ सामने आती हैं:
(i) हजारों जीन में सीमित GTFs के लिए संघर्ष होता है ।
(ii) कुछ प्रमोटर्स में जरूरी क्षारक क्रम में हुए फेरबदल के कारण, डी. एन. ए. बाइण्डिंग प्रोटीन्स से जुड़ने की क्षमता कम हो जाती है ।
(iii) चूँकि DNA क्रोमेटिन के रूप में पैक रहता है । अत: GTFs से प्रमोटर तक पहुंचने में कठिनाई होती है ।
(iv) कुछ प्रोटीन कारक प्रारम्भिक जटिल (Initiation Complex) के निर्माण में रुकावट उत्पन्न करते हैं ।
आर. एन. ए. स्प्लाइसिंग (RNA Splicing):
यूकैरियोटिक जीवधारियों में परिपक्व RNA प्राप्त करने के लिए RNA के प्राथमिक अनुलेख की स्प्लाइसिंग अत्यन्त आवश्यक होती है । स्प्लाइसिंग (Splicing) की इस प्रक्रिया में आर. एन. ए. के निरर्थक खण्डों, (इण्ट्रॉन द्वारा कोडित) जो किसी प्रोटीन को कोडित नहीं करते को निकल दिया जाता है । ज्ञातव्य है कि स्प्लिट जीन्स (Split Genes) में एक्जॉन खण्डों के बीच-बीच में इण्ट्रान्स भी पाये जाते हैं ।
स्प्लाइसिंग प्रक्रिया मुख्यतः निम्न प्रकार से सम्पन्न हो सकती है:
(i) कवक माइटोकॉण्ड्रीया (Fungal Mitochondria) व अन्य समूह 1 के इण्ट्रॉन्स की स्वतः स्प्लाइसिंग ।
(ii) यूकैरियोट्स के न्यूक्लियर इण्ट्रॉन्स की स्प्लाइसिंग- इस प्रकार की स्प्लाइसिंग में एक्जॉन-इण्ट्रॉन जंक्शन पर स्प्लाइसोम बनता है ।